
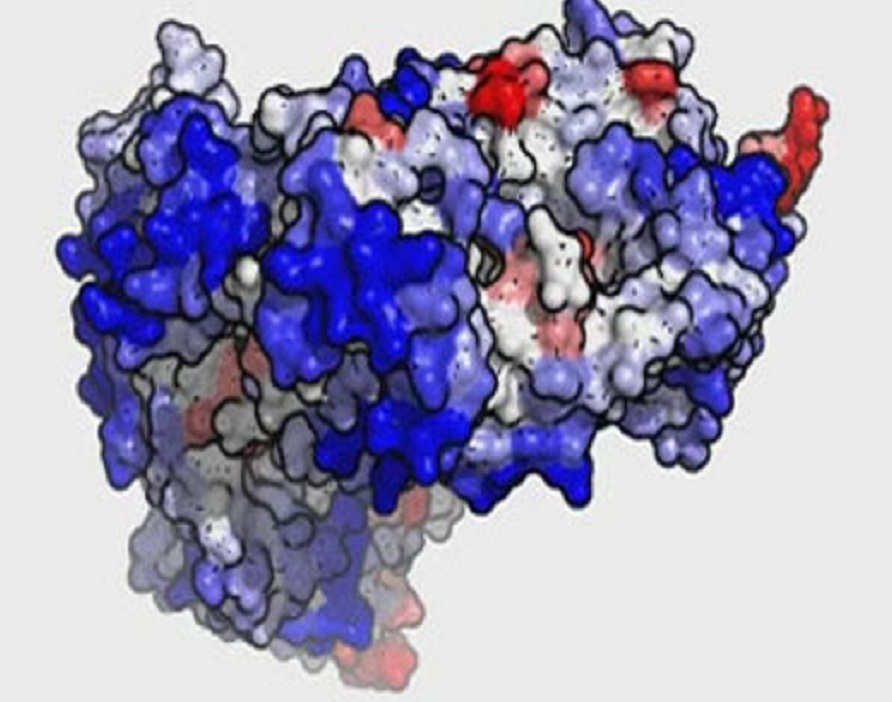
Modelado del anticuerpo monoclonal Rituximab, empleado en el tratamiento de linfomas y leucemia. Las zonas rojas corresponden a las regiones con más propensión al agregamiento, que podrían ser rediseñadas con el Aggrescan3D para mejorar la funcionalidad del fármaco. Fuente: UAB.
Investigadores del Instituto de Biotecnología y Biomedicina (IBB) de la Universidad Autónoma de Barcelona y de la Universidad de Varsovia (Polonia) han desarrollado Aggrescan3D, un nuevo método computacional que permite estudiar la estructura de las proteínas globulares plegadas y su propensión a formar agregados proteicos tóxicos.
El conocimiento actual de las bases moleculares de la agregación proteica, causante de numerosas patologías, ha generado una serie de algoritmos para identificar las regiones de las proteínas propensas a agregarse. Entre ellos, el Aggrescan, desarrollado hace ocho años por los mismos investigadores del IBB, que fue uno de los primeros métodos computacionales que se crearon a tal fin.
Pero la mayoría de estos algoritmos analiza sólo las regiones que están en la secuencia lineal de las proteínas. Esto dificulta predecir las propiedades de agregación de las proteínas globulares, donde estas secuencias están a menudo protegidas dentro de su estructura esférica nativa.
El Aggrescan3D (A3D), que se ha implementado como un servidor web de libre acceso para el mundo académico, supera estas limitaciones con una aproximación basada en la estructura de las proteínas desde estados plegados. Según el artículo en el que lo presentan los investigadores, publicado en Nucleic Acids Research, el nuevo algoritmo tiene una precisión significativamente más alta que los basados en secuencias lineales para predecir las propiedades de agregación de las proteínas globulares y aporta nuevas e importantes prestaciones, entre las que destaca la posibilidad de modelar fácilmente mutaciones patogénicas o rediseñar proteínas de interés terapéutico, como anticuerpos, con funcionalidad incrementada.
“Hasta el momento, el A3D es el algoritmo más rápido disponible para predecir la agregación de proteínas que puede trabajar en un modo dinámico, esto es, teniendo en cuenta la flexibilidad de la estructura de la proteína. Esto nos permite modelar agregaciones atribuibles a las fluctuaciones naturales de la estructura, así como las que están causadas por mutaciones patogénicas desestabilizadoras y predecir su impacto en la propensión a la agregación”, señala en la nota de prensa de la universidad Salvador Ventura, investigador del IBB y del Departamento de Bioquímica y Biología Molecular de la UAB, que ha coordinado la creación del nuevo método.
El conocimiento actual de las bases moleculares de la agregación proteica, causante de numerosas patologías, ha generado una serie de algoritmos para identificar las regiones de las proteínas propensas a agregarse. Entre ellos, el Aggrescan, desarrollado hace ocho años por los mismos investigadores del IBB, que fue uno de los primeros métodos computacionales que se crearon a tal fin.
Pero la mayoría de estos algoritmos analiza sólo las regiones que están en la secuencia lineal de las proteínas. Esto dificulta predecir las propiedades de agregación de las proteínas globulares, donde estas secuencias están a menudo protegidas dentro de su estructura esférica nativa.
El Aggrescan3D (A3D), que se ha implementado como un servidor web de libre acceso para el mundo académico, supera estas limitaciones con una aproximación basada en la estructura de las proteínas desde estados plegados. Según el artículo en el que lo presentan los investigadores, publicado en Nucleic Acids Research, el nuevo algoritmo tiene una precisión significativamente más alta que los basados en secuencias lineales para predecir las propiedades de agregación de las proteínas globulares y aporta nuevas e importantes prestaciones, entre las que destaca la posibilidad de modelar fácilmente mutaciones patogénicas o rediseñar proteínas de interés terapéutico, como anticuerpos, con funcionalidad incrementada.
“Hasta el momento, el A3D es el algoritmo más rápido disponible para predecir la agregación de proteínas que puede trabajar en un modo dinámico, esto es, teniendo en cuenta la flexibilidad de la estructura de la proteína. Esto nos permite modelar agregaciones atribuibles a las fluctuaciones naturales de la estructura, así como las que están causadas por mutaciones patogénicas desestabilizadoras y predecir su impacto en la propensión a la agregación”, señala en la nota de prensa de la universidad Salvador Ventura, investigador del IBB y del Departamento de Bioquímica y Biología Molecular de la UAB, que ha coordinado la creación del nuevo método.
Una cuestión clave
El nuevo algoritmo se puede aplicar en cualquier proteína de la cual se conoce la estructura o que pueda ser generada por modelización. Para validar el nuevo método, los investigadores han usado proteínas con propiedad de agregación caracterizadas ya experimentalmente. En el modo estático, se pueden estudiar proteínas individuales o complejos proteicos de hasta 20.000 átomos, y en el modo dinámico, proteínas de hasta 400 aminoácidos.
La agregación proteica ha pasado de ser un área ignorada de la química de proteínas a una cuestión clave en biomedicina y biotecnología.
“El mal plegamiento de las proteínas y la agregación subsecuente está detrás de un número creciente de enfermedades humanas, como la enfermedad de Alzheimer, el Párkinson o la Diabetes Tipo II, y es una de las barreras más importantes para diseñar y fabricar proteínas para aplicaciones terapéuticas. Estas terapias, que implican el uso de anticuerpos monoclonales, factores de crecimiento o la sustitución de enzimas, han demostrado ya tener una alta precisión hacia sus dianas moleculares, por ello profundizar en su estudio resulta tan trascendente”, concluye Ventura.
Hace unos meses, científicos del Institute for Research on Biomedecine (IRB) de Barcelona, en colaboración con investigadores de la Universidad de Barcelona, observaron que una de estas agregaciones, en concreto de agrupaciones de entre 20 a 100 unidades del péptido beta amiloide, asociado al Alzheimer, las hace nocivas para las neuronas.
El nuevo algoritmo se puede aplicar en cualquier proteína de la cual se conoce la estructura o que pueda ser generada por modelización. Para validar el nuevo método, los investigadores han usado proteínas con propiedad de agregación caracterizadas ya experimentalmente. En el modo estático, se pueden estudiar proteínas individuales o complejos proteicos de hasta 20.000 átomos, y en el modo dinámico, proteínas de hasta 400 aminoácidos.
La agregación proteica ha pasado de ser un área ignorada de la química de proteínas a una cuestión clave en biomedicina y biotecnología.
“El mal plegamiento de las proteínas y la agregación subsecuente está detrás de un número creciente de enfermedades humanas, como la enfermedad de Alzheimer, el Párkinson o la Diabetes Tipo II, y es una de las barreras más importantes para diseñar y fabricar proteínas para aplicaciones terapéuticas. Estas terapias, que implican el uso de anticuerpos monoclonales, factores de crecimiento o la sustitución de enzimas, han demostrado ya tener una alta precisión hacia sus dianas moleculares, por ello profundizar en su estudio resulta tan trascendente”, concluye Ventura.
Hace unos meses, científicos del Institute for Research on Biomedecine (IRB) de Barcelona, en colaboración con investigadores de la Universidad de Barcelona, observaron que una de estas agregaciones, en concreto de agrupaciones de entre 20 a 100 unidades del péptido beta amiloide, asociado al Alzheimer, las hace nocivas para las neuronas.
Referencia bibliográfica:
Rafael Zambrano, Michal Jamroz, Agata Szczasiuk, Jordi Pujols,Sebastian Kmiecik y Salvador Ventura. AGGRESCAN3D (A3D): server for prediction of aggregation properties of protein structures. Nucleic Acids Research (2015). DOI: 10.1093/nar/gkv359.
Rafael Zambrano, Michal Jamroz, Agata Szczasiuk, Jordi Pujols,Sebastian Kmiecik y Salvador Ventura. AGGRESCAN3D (A3D): server for prediction of aggregation properties of protein structures. Nucleic Acids Research (2015). DOI: 10.1093/nar/gkv359.

 Tendencias Científicas
Tendencias Científicas

 La diversidad intestinal de los bebés los protege de asma y alergias futuras
La diversidad intestinal de los bebés los protege de asma y alergias futuras CIENCIA ON LINE
CIENCIA ON LINE