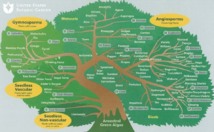
Árbol botánico de la vida. Fuente: opentreeoflife.wordpress.com.
Con la publicación de su obra fundamental en 1859, El origen de las especies, Charles Darwin sentó las bases sobre cómo los seres vivos han evolucionado con el tiempo a partir de un antepasado común, mediante un proceso denominado selección natural. Aquel libro contenía únicamente una ilustración, una representación de un árbol descendiente conocido como Árbol de la Vida.
Durante más de 150 años, científicos de todo el mundo han continuado construyendo árboles evolutivos, también llamados filogenéticos, a su imagen y semejanza. Su famosa Teoría de la Evolución brindó a biólogos y paleontólogos el enorme desafío de descubrir el patrón de ramificación del árbol. Sin embargo, a pesar de significativos progresos para dar más cuerpo a las ramas principales, aún no existe un espacio común donde todos los investigadores puedan acudir para visualizar y analizar el Árbol de la Vida en toda su magnitud.
Ese es el reto al que se enfrentan ahora tres equipos de científicos unidos por el programa AVAToL -resultado de las siglas en inglés de Montar, Visualizar y Analizar el Árbol de la Vida-, un proyecto impulsado por la Fundación Nacional de Ciencia (NSF) de Estados Unidos mediante una subvención de 13 millones de euros.
Como explica la propia agencia de investigación norteamericana en un comunicado, el objetivo es crear una exhaustiva estructura donde tenga cabida todo el conocimiento que los científicos han revelado hasta el momento sobre cómo se relacionan las diferentes especies, desde la bacteria más pequeña al árbol más alto.
“El programa AVAToL es una nueva dirección emocionante en el área de la biología”, señala Alan Townsend, director de la división de Biología Ambiental de la NSF. “Es fundamental para comprender la relación cambiante entre la sociedad humana y la biodiversidad de la Tierra”, añade.
Durante más de 150 años, científicos de todo el mundo han continuado construyendo árboles evolutivos, también llamados filogenéticos, a su imagen y semejanza. Su famosa Teoría de la Evolución brindó a biólogos y paleontólogos el enorme desafío de descubrir el patrón de ramificación del árbol. Sin embargo, a pesar de significativos progresos para dar más cuerpo a las ramas principales, aún no existe un espacio común donde todos los investigadores puedan acudir para visualizar y analizar el Árbol de la Vida en toda su magnitud.
Ese es el reto al que se enfrentan ahora tres equipos de científicos unidos por el programa AVAToL -resultado de las siglas en inglés de Montar, Visualizar y Analizar el Árbol de la Vida-, un proyecto impulsado por la Fundación Nacional de Ciencia (NSF) de Estados Unidos mediante una subvención de 13 millones de euros.
Como explica la propia agencia de investigación norteamericana en un comunicado, el objetivo es crear una exhaustiva estructura donde tenga cabida todo el conocimiento que los científicos han revelado hasta el momento sobre cómo se relacionan las diferentes especies, desde la bacteria más pequeña al árbol más alto.
“El programa AVAToL es una nueva dirección emocionante en el área de la biología”, señala Alan Townsend, director de la división de Biología Ambiental de la NSF. “Es fundamental para comprender la relación cambiante entre la sociedad humana y la biodiversidad de la Tierra”, añade.
Proyectos anteriores
Los investigadores están trabajando para proporcionar la infraestructura que requiere el desarrollo de un Árbol de la Vida virtual, así como las herramientas informáticas necesarias para su posterior análisis y visualización en todo el mundo. Estas son las claves que diferenciarán esta estructura de las creadas hasta el momento.
Y es que no será por falta de investigación o de datos. Los avances en la secuenciación del ADN y el análisis de la evolución, el descubrimiento de fósiles primitivos fundamentales, o los nuevos métodos y herramientas dan lugar cada año a la publicación de miles de nuevos árboles evolutivos en revistas científicas. Sin embargo, la mayoría de ellos se centra en ramas específicas y desconectadas del Árbol de la Vida.
Parte de la dificultad de unir toda esa información radica en la propia grandeza de la tarea. Los árboles evolutivos más grandes hasta la fecha contienen alrededor de 100.000 grupos de organismos. Montar las ramas de todas las especies de animales, plantas, hongos y microbios -y el recuento de las que aún quedan por nombrar o descubrir- requiriere de nuevas herramientas informáticas capaces de analizar, combinar y conectar grandes cantidades de datos procedentes de árboles ya publicados en un todo sintético.
Otra dificultad radica en cómo los científicos difunden generalmente sus resultados. Sólo una pequeña fracción de todos los árboles evolutivos generados ha llegado a publicarse, de la cual los investigadores estiman que un mero cuatro por ciento termina en una base de datos en formato digital. Así, “la mayor parte del conocimiento está encerrado en estáticos artículos de revistas, en formatos de archivo que pueden ser difíciles de descargar, volver a analizar o fusionar con nueva información por parte de otros investigadores”, destacan desde la NSF.
AVAToL pretende romper con la tradición. Precisamente lo que diferencia este programa de esfuerzos anteriores, según los científicos, es su alcance, su enfoque hacia la creación de una estructura abierta, dinámica y evolutiva, capaz de perfeccionarse continuamente mientras se recopilan nuevos datos de la biodiversidad, y se desarrollan herramientas informáticas para ampliar los análisis evolutivos basados en el árbol.
Los investigadores están trabajando para proporcionar la infraestructura que requiere el desarrollo de un Árbol de la Vida virtual, así como las herramientas informáticas necesarias para su posterior análisis y visualización en todo el mundo. Estas son las claves que diferenciarán esta estructura de las creadas hasta el momento.
Y es que no será por falta de investigación o de datos. Los avances en la secuenciación del ADN y el análisis de la evolución, el descubrimiento de fósiles primitivos fundamentales, o los nuevos métodos y herramientas dan lugar cada año a la publicación de miles de nuevos árboles evolutivos en revistas científicas. Sin embargo, la mayoría de ellos se centra en ramas específicas y desconectadas del Árbol de la Vida.
Parte de la dificultad de unir toda esa información radica en la propia grandeza de la tarea. Los árboles evolutivos más grandes hasta la fecha contienen alrededor de 100.000 grupos de organismos. Montar las ramas de todas las especies de animales, plantas, hongos y microbios -y el recuento de las que aún quedan por nombrar o descubrir- requiriere de nuevas herramientas informáticas capaces de analizar, combinar y conectar grandes cantidades de datos procedentes de árboles ya publicados en un todo sintético.
Otra dificultad radica en cómo los científicos difunden generalmente sus resultados. Sólo una pequeña fracción de todos los árboles evolutivos generados ha llegado a publicarse, de la cual los investigadores estiman que un mero cuatro por ciento termina en una base de datos en formato digital. Así, “la mayor parte del conocimiento está encerrado en estáticos artículos de revistas, en formatos de archivo que pueden ser difíciles de descargar, volver a analizar o fusionar con nueva información por parte de otros investigadores”, destacan desde la NSF.
AVAToL pretende romper con la tradición. Precisamente lo que diferencia este programa de esfuerzos anteriores, según los científicos, es su alcance, su enfoque hacia la creación de una estructura abierta, dinámica y evolutiva, capaz de perfeccionarse continuamente mientras se recopilan nuevos datos de la biodiversidad, y se desarrollan herramientas informáticas para ampliar los análisis evolutivos basados en el árbol.
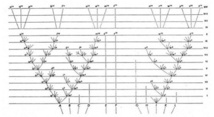
Diagrama dibujado por Charles Darwin en El origen de las especies. Fuente: Wikimedia Commons.
Tres equipos
Para su consecución, el proyecto consta de tres partes perfectamente diferenciadas y asignadas a equipos de investigación distintos. El primero de ellos, dirigido por la investigadora Karen Cranston, de la Universidad de Duke y el Centro Nacional de Síntesis Evolutiva, ambos en Carolina del Norte (EEUU), será el encargado de crear el árbol online con los 1,8 millones de especies nombradas, accesible tanto para los científicos como el público en general.
El montaje del árbol lleva aparejada la incorporación previa de todos los resultados publicados y el esfuerzo por desarrollar, probar y mejorar métodos de síntesis de datos. Esta estructura inicial, conocida como Árbol Abierto de la Vida, no será estática, sino que podrá actualizarse y revisarse conforme aparezcan nuevos datos. Los investigadores pondrán incluso descargarla para ampliar sus estudios.
El segundo grupo, pilotado por Lucas Harmon, de la Universidad de Idaho, se enfrentará al reto de organizar la información en un formato manejable, donde los científicos puedan ver fácilmente cómo se relacionan los organismos, y que al mismo tiempo sea capaz de propiciar nuevos conocimientos científicos en todas las áreas de la biología comparada.
Y es que descubrir cómo están relacionados entre sí los millones de especies de la Tierra no sólo es importante para localizar los parientes más cercanos de un antílope, o determinar si el atún está más estrechamente ligado a la estrella de mar o la lamprea. Las relaciones evolutivas pueden ayudar a los expertos en biología comparada a identificar nuevos y prometedores medicamentos, a conseguir desarrollos más resistentes, cultivos de mayor rendimiento, o a luchar contra enfermedades infecciosas como el VIH, el ántrax o la gripe.
Por último el tercer equipo, encabezado por Maureen O’Leary, de la Universidad de Stony Brook, en Nueva York (EEUU), está formado por biólogos, informáticos y paleontólogos encargados de extender y adaptar los avances más recientes en análisis de imágenes, aprendizaje automático y procesamiento del lenguaje natural para permitir un estudio rápido y automatizado de los fenotipos de las especies a gran escala a través del Árbol de la Vida.
La fenómica es un área de la biología que mide cómo cambian los rasgos físicos y bioquímicos de organismos en respuesta a mutaciones genéticas e influencias ambientales. Los fenotipos permitirán a los científicos reconstruir la historia evolutiva de las especies fósiles, a su vez, crucial para la comprensión de la historia de la vida.
Para su consecución, el proyecto consta de tres partes perfectamente diferenciadas y asignadas a equipos de investigación distintos. El primero de ellos, dirigido por la investigadora Karen Cranston, de la Universidad de Duke y el Centro Nacional de Síntesis Evolutiva, ambos en Carolina del Norte (EEUU), será el encargado de crear el árbol online con los 1,8 millones de especies nombradas, accesible tanto para los científicos como el público en general.
El montaje del árbol lleva aparejada la incorporación previa de todos los resultados publicados y el esfuerzo por desarrollar, probar y mejorar métodos de síntesis de datos. Esta estructura inicial, conocida como Árbol Abierto de la Vida, no será estática, sino que podrá actualizarse y revisarse conforme aparezcan nuevos datos. Los investigadores pondrán incluso descargarla para ampliar sus estudios.
El segundo grupo, pilotado por Lucas Harmon, de la Universidad de Idaho, se enfrentará al reto de organizar la información en un formato manejable, donde los científicos puedan ver fácilmente cómo se relacionan los organismos, y que al mismo tiempo sea capaz de propiciar nuevos conocimientos científicos en todas las áreas de la biología comparada.
Y es que descubrir cómo están relacionados entre sí los millones de especies de la Tierra no sólo es importante para localizar los parientes más cercanos de un antílope, o determinar si el atún está más estrechamente ligado a la estrella de mar o la lamprea. Las relaciones evolutivas pueden ayudar a los expertos en biología comparada a identificar nuevos y prometedores medicamentos, a conseguir desarrollos más resistentes, cultivos de mayor rendimiento, o a luchar contra enfermedades infecciosas como el VIH, el ántrax o la gripe.
Por último el tercer equipo, encabezado por Maureen O’Leary, de la Universidad de Stony Brook, en Nueva York (EEUU), está formado por biólogos, informáticos y paleontólogos encargados de extender y adaptar los avances más recientes en análisis de imágenes, aprendizaje automático y procesamiento del lenguaje natural para permitir un estudio rápido y automatizado de los fenotipos de las especies a gran escala a través del Árbol de la Vida.
La fenómica es un área de la biología que mide cómo cambian los rasgos físicos y bioquímicos de organismos en respuesta a mutaciones genéticas e influencias ambientales. Los fenotipos permitirán a los científicos reconstruir la historia evolutiva de las especies fósiles, a su vez, crucial para la comprensión de la historia de la vida.

 Tendencias Científicas
Tendencias Científicas

 La Inteligencia Artificial habla sobre la legalización del cannabis
La Inteligencia Artificial habla sobre la legalización del cannabis CIENCIA ON LINE
CIENCIA ON LINE