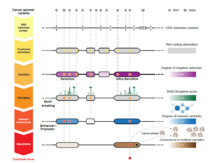
Funcionamiento del método computacional de búsqueda de ADN no codificante 'ultrasensible'. Fuente: Sanger Institute/Science.
Investigadores de varios centros británicos han conseguido identificar regiones de ADN dentro del ADN no codificante, la parte principal del genoma que no se traduce en proteínas, donde las mutaciones pueden causar enfermedades como el cáncer .
Su enfoque, publicado en la revista Science, revela muchas variantes genéticas potenciales dentro del ADN no codificante que impulsan el desarrollo de diferentes tipos de cáncer, y abre la posibilidad de encontrar mutaciones que provoquen otro tipo de enfermedades.
A diferencia de la región codificante del genoma, donde se encuentran los 23.000 genes codificadores de proteínas, la región no codificante -que conforma el 98 % de nuestro genoma- es poco conocida. Estudios recientes han puesto de relieve el valor biológico de las regiones no codificantes, que antes se consideraban ADN "basura", en la regulación de las proteínas. Esta nueva información proporciona un punto de partida para los investigadores para recorrer las regiones no codificantes e identificar las regiones funcionalmente importantes.
"Nuestra técnica permite a los científicos centrarse en las partes más funcionalmente importantes de las regiones no codificantes del genoma", explica el profesor Mark Gerstein, autor principal del artículo, de la Universidad de Yale. "Esto no es sólo beneficioso para la investigación del cáncer, sino que también se puede extender a otras enfermedades genéticas", señala en la nota de prensa publicada por el Wellcome Trust Sanger Institute.de Cambridge.
El equipo utilizó el conjunto de variantes genéticas de la primera fase del Proyecto 1000 Genomas, junto con información sobre las regiones no codificantes generadas por el proyecto Encode, y encontró regiones que no presentaban mucha variación. Los genes que codifican proteínas juegan un papel crucial en la supervivencia y la salud del ser humano, y sufren una fuerte selección purificadora, que elimina la variabilidad. El equipo encontró que algunas regiones de ADN no codificante mostraron casi los mismos bajos niveles de variación como los genes que codifican proteínas, y llamó a estas regiones ultrasensibles.
Dentro de las regiones ultrasensibles, observaron las letras de ADN que, cuando eran alteradas, causaban una mayor perturbación en la región genética. Si esta región no codificante y ultrasensible es básica para una red de muchos genes relacionados, una variación en ell puede causar un mayor efecto en cadena, que provoque una enfermedad.
El método obtenido
Los investigadores juntaron toda esta información para desarrollar un proceso de trabajo computacional conocido como FunSeq. Este sistema da prioridad a las variantes genéticas en las regiones no codificantes en función de su impacto previsto sobre las enfermedades humanas.
Su enfoque, publicado en la revista Science, revela muchas variantes genéticas potenciales dentro del ADN no codificante que impulsan el desarrollo de diferentes tipos de cáncer, y abre la posibilidad de encontrar mutaciones que provoquen otro tipo de enfermedades.
A diferencia de la región codificante del genoma, donde se encuentran los 23.000 genes codificadores de proteínas, la región no codificante -que conforma el 98 % de nuestro genoma- es poco conocida. Estudios recientes han puesto de relieve el valor biológico de las regiones no codificantes, que antes se consideraban ADN "basura", en la regulación de las proteínas. Esta nueva información proporciona un punto de partida para los investigadores para recorrer las regiones no codificantes e identificar las regiones funcionalmente importantes.
"Nuestra técnica permite a los científicos centrarse en las partes más funcionalmente importantes de las regiones no codificantes del genoma", explica el profesor Mark Gerstein, autor principal del artículo, de la Universidad de Yale. "Esto no es sólo beneficioso para la investigación del cáncer, sino que también se puede extender a otras enfermedades genéticas", señala en la nota de prensa publicada por el Wellcome Trust Sanger Institute.de Cambridge.
El equipo utilizó el conjunto de variantes genéticas de la primera fase del Proyecto 1000 Genomas, junto con información sobre las regiones no codificantes generadas por el proyecto Encode, y encontró regiones que no presentaban mucha variación. Los genes que codifican proteínas juegan un papel crucial en la supervivencia y la salud del ser humano, y sufren una fuerte selección purificadora, que elimina la variabilidad. El equipo encontró que algunas regiones de ADN no codificante mostraron casi los mismos bajos niveles de variación como los genes que codifican proteínas, y llamó a estas regiones ultrasensibles.
Dentro de las regiones ultrasensibles, observaron las letras de ADN que, cuando eran alteradas, causaban una mayor perturbación en la región genética. Si esta región no codificante y ultrasensible es básica para una red de muchos genes relacionados, una variación en ell puede causar un mayor efecto en cadena, que provoque una enfermedad.
El método obtenido
Los investigadores juntaron toda esta información para desarrollar un proceso de trabajo computacional conocido como FunSeq. Este sistema da prioridad a las variantes genéticas en las regiones no codificantes en función de su impacto previsto sobre las enfermedades humanas.
"Nuestro método es una forma práctica de buscar selección purificadora en regiones no codificantes del genoma a partir de datos de libre acceso, como los de Encode y 1000 Genomas", expica Yali Xue, del Wellcome Trust Sanger Institute. "Realmente muestra el valor de estos conjuntos a gran escala de datos de acceso abierto."
El equipo aplicó FunSeq a 90 genomas de cáncer, incluyendo cáncer de mama, cáncer de próstata y tumores cerebrales, y encontró cerca de 100 variantes posibles de ADN no codificante relacionadas con el cáncer. En los genomas del cáncer de mama, por ejemplo, se encontraron que un solo cambio de letra de ADN parece tener un gran impacto en su desarrollo. Este cambio de una sola letra se produce en una región ultrasensible que es central para una red de muchos genes relacionados . "Este método puede aplicarse a todo tipo de enfermedades", añade Chris Tyler-Smith, del Sanger Institute.
Otra investigación
Por su parte, investigadores de la Universidad de Montreal han descubierto que las moléculas de telomerasa, esenciales para el desarrollo del cáncer, se unen entre sí gracias a una molécula denominada Terra, de tipo ARN no codificante, cuya utilidad se desconocía. El ARN no codificantes constituye la "materia oscura del genoma", como la definen los investigadores.
A medida que las células se dividen, los cromosomas, las largas cadenas de moléculas de ADN que codifican los genes, deben duplicarse. En el proceso, los telómeros, que informan al sistema de duplicación de dónde está el final de los cromosomas, se van acortando. La telomerasa hace que recuperen su longitud, pero en la mayoría de células está inactiva, lo cual acaba produciendo la desaparición de los telómeros y que las células dejen de duplicarse, y mueran. En cambio, en las del cáncer sí funciona, lo que hace que las células cancerosas sean inmortales.
El gen de Terra se había encontrado ya en los telómeros y se sospechaba que la molécula jugaban un papel importante en la integridad de éstos. Los investigadores descubrieron que la producción de Terra se activaba cuando el telómero en el que está su gen relacionado se acorta. A continuación, descubrieron que las moléculas de Terra juntan en un solo lugar y al mismo tiempo las moléculas de la telomerasa que han recopilado, que después son dirigidas al telómero en el que se originó la propia molécula de Terra.
El equipo aplicó FunSeq a 90 genomas de cáncer, incluyendo cáncer de mama, cáncer de próstata y tumores cerebrales, y encontró cerca de 100 variantes posibles de ADN no codificante relacionadas con el cáncer. En los genomas del cáncer de mama, por ejemplo, se encontraron que un solo cambio de letra de ADN parece tener un gran impacto en su desarrollo. Este cambio de una sola letra se produce en una región ultrasensible que es central para una red de muchos genes relacionados . "Este método puede aplicarse a todo tipo de enfermedades", añade Chris Tyler-Smith, del Sanger Institute.
Otra investigación
Por su parte, investigadores de la Universidad de Montreal han descubierto que las moléculas de telomerasa, esenciales para el desarrollo del cáncer, se unen entre sí gracias a una molécula denominada Terra, de tipo ARN no codificante, cuya utilidad se desconocía. El ARN no codificantes constituye la "materia oscura del genoma", como la definen los investigadores.
A medida que las células se dividen, los cromosomas, las largas cadenas de moléculas de ADN que codifican los genes, deben duplicarse. En el proceso, los telómeros, que informan al sistema de duplicación de dónde está el final de los cromosomas, se van acortando. La telomerasa hace que recuperen su longitud, pero en la mayoría de células está inactiva, lo cual acaba produciendo la desaparición de los telómeros y que las células dejen de duplicarse, y mueran. En cambio, en las del cáncer sí funciona, lo que hace que las células cancerosas sean inmortales.
El gen de Terra se había encontrado ya en los telómeros y se sospechaba que la molécula jugaban un papel importante en la integridad de éstos. Los investigadores descubrieron que la producción de Terra se activaba cuando el telómero en el que está su gen relacionado se acorta. A continuación, descubrieron que las moléculas de Terra juntan en un solo lugar y al mismo tiempo las moléculas de la telomerasa que han recopilado, que después son dirigidas al telómero en el que se originó la propia molécula de Terra.
Referencia bibliográfica:
Ekta Khurana, Yao Fu, Vincenza Colonna, Xinmeng Jasmine Mu et al.: Integrative annotation of variants from 1,092 humans: application to cancer genomics. Science 2013 DOI: 10.1126/science.1235587
Emilio Cusanelli, Carmina Angelica Perez Romero, Pascal Chartrand. Telomeric Noncoding RNA TERRA Is Induced by Telomere Shortening to Nucleate Telomerase Molecules at Short Telomeres. Molecular Cell, 26 September 2013 (Vol. 51, Issue 6, pp. 780-791)
Ekta Khurana, Yao Fu, Vincenza Colonna, Xinmeng Jasmine Mu et al.: Integrative annotation of variants from 1,092 humans: application to cancer genomics. Science 2013 DOI: 10.1126/science.1235587
Emilio Cusanelli, Carmina Angelica Perez Romero, Pascal Chartrand. Telomeric Noncoding RNA TERRA Is Induced by Telomere Shortening to Nucleate Telomerase Molecules at Short Telomeres. Molecular Cell, 26 September 2013 (Vol. 51, Issue 6, pp. 780-791)

 Tendencias Científicas
Tendencias Científicas

 El corazón débil también daña al cerebro
El corazón débil también daña al cerebro CIENCIA ON LINE
CIENCIA ON LINE