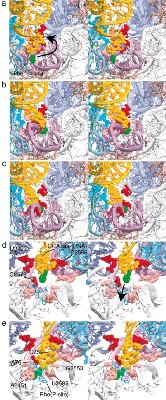
Los ribosomas, observador desde la primera fila.
Investigadores de Los Alamos National Laboratory, en Estados Unidos, han desarrollado un simulador informático que reproduce los procesos de los ribosomas. Tal como explica el citado laboratorio en un comunicado, se trata de un programa de ordenador (la “Q Machine") que reproduce los procesos dinámicos de más de dos millones y medio de átomos en movimiento. Esta reproducción es seis veces mayor que cualquier otra simulación producida hasta la fecha. La simulación ha sido realizada a escala atómica.
Los ribosomas son orgánulos que se encuentran en todas las células vivas (menos en los espermatozoides) y se encargan de la síntesis de proteínas. Su función consiste en ensamblar las proteínas siguiendo la información genética que reciben del ADN (en forma de ARN mensajero). La síntesis proteica de las células es llevada a cabo por los ribosomas gracias a dos subunidades del ribosoma que se encajan y trabajan en conjunción para la traducción del ARN mensajero (ARN m).
Hasta ahora, las simulaciones llevadas a cabo de los ribosomas habían sido estáticas. El movimiento permitirá conocer mejor su funcionamiento, con el fin de desarrollar antibióticos más eficaces para enfermedades tan letales como el ántrax.
La trascripción del ADN, clave para la vida
La traducción de la información que llega del ADN para realizar la síntesis proteica de las células es clave para la supervivencia de todos los organismos terrestres. Aunque esta “decodificación” o trascripción del ARN m había sido estudiada durante más de cuatro décadas, hasta hace muy poco no se había identificado al completo la forma en que los ribosomas hacen la selección.
Según publica la revista Proceedings of the National Academy of Sciences, la simulación muestra que existe una galería dentro del ribosoma que la transferencia de ARN m debe atravesar, con el fin de que la trascripción de la información genética se realice. Esta galería parece estar fabricada casi al completo con principios básicos universales y es una región del ribosoma que contiene una variedad de novedosos objetivos para los antibióticos.
Asimismo, la simulación también revela que la molécula del ARN m, esencial para la trascripción de la información genética de la síntesis proteica, debe ser adaptable en dos de sus partes, con el fin de que la decodificación se dé durante el proceso de acoplamiento entre el ARN m y los ribosomas.
De la simulación se desprende información que servirá tanto para el futuro desarrollo de máquinas moleculares biológicas y artificiales como para próximas investigaciones en el campo de la bioquímica, centradas en la identificación de una veintena de principios básicos que componen el ribosoma, y que son imprescindibles para el proceso de acoplamiento de ribosomas y ARN m. Asimismo, se podrán conocer mejor las pautas estructurales que actúan en los mecanismos de selección del ARN por parte de los ribosomas.
Problemas superados
Por último, esta nueva técnica ofrece una novedosa y poderosa herramienta para comprender mejor las máquinas moleculares –lo que permitirá desarrollar máquinas biológicas y artificiales a escala nanométrica-, así como para aumentar la eficacia de los antibióticos, que dentro de la maquinaria de la célula podrían comportarse como una especie de llave mecánica capaz de detener determinados procesos celulares dañinos para el organismo.
El diseño de medicamentos basados en la reproducción o simulación sólo de las estructuras estáticas del ribosoma podría compararse con el intento de interceptar en pleno vuelo un misil, si contáramos con la única información de su lugar de lanzamiento y su objetivo. Así, resulta imposible averiguar la trayectoria exacta del misil, del mismo modo que resulta muy difícil conocer los pasos que siguen los ribosomas en sus procesos si no existe una simulación dinámica de éstos.
La reproducción en movimiento de la actividad de los ribosomas ha sido posible gracias a la combinación de diversas ramas de la ciencia: la bioquímica, la informática, la biología molecular, la física, la biología estructural y las ciencias materiales. El resultado permitirá conocer mejor los procesamientos de información a escala nanométrica, tanto artificiales como biológicos. Y es que, de hecho, el ribosoma no es otra cosa que un ordenador a nano escala, un procesador de datos que trabaja para el funcionamiento de la célula.
Los ribosomas son orgánulos que se encuentran en todas las células vivas (menos en los espermatozoides) y se encargan de la síntesis de proteínas. Su función consiste en ensamblar las proteínas siguiendo la información genética que reciben del ADN (en forma de ARN mensajero). La síntesis proteica de las células es llevada a cabo por los ribosomas gracias a dos subunidades del ribosoma que se encajan y trabajan en conjunción para la traducción del ARN mensajero (ARN m).
Hasta ahora, las simulaciones llevadas a cabo de los ribosomas habían sido estáticas. El movimiento permitirá conocer mejor su funcionamiento, con el fin de desarrollar antibióticos más eficaces para enfermedades tan letales como el ántrax.
La trascripción del ADN, clave para la vida
La traducción de la información que llega del ADN para realizar la síntesis proteica de las células es clave para la supervivencia de todos los organismos terrestres. Aunque esta “decodificación” o trascripción del ARN m había sido estudiada durante más de cuatro décadas, hasta hace muy poco no se había identificado al completo la forma en que los ribosomas hacen la selección.
Según publica la revista Proceedings of the National Academy of Sciences, la simulación muestra que existe una galería dentro del ribosoma que la transferencia de ARN m debe atravesar, con el fin de que la trascripción de la información genética se realice. Esta galería parece estar fabricada casi al completo con principios básicos universales y es una región del ribosoma que contiene una variedad de novedosos objetivos para los antibióticos.
Asimismo, la simulación también revela que la molécula del ARN m, esencial para la trascripción de la información genética de la síntesis proteica, debe ser adaptable en dos de sus partes, con el fin de que la decodificación se dé durante el proceso de acoplamiento entre el ARN m y los ribosomas.
De la simulación se desprende información que servirá tanto para el futuro desarrollo de máquinas moleculares biológicas y artificiales como para próximas investigaciones en el campo de la bioquímica, centradas en la identificación de una veintena de principios básicos que componen el ribosoma, y que son imprescindibles para el proceso de acoplamiento de ribosomas y ARN m. Asimismo, se podrán conocer mejor las pautas estructurales que actúan en los mecanismos de selección del ARN por parte de los ribosomas.
Problemas superados
Por último, esta nueva técnica ofrece una novedosa y poderosa herramienta para comprender mejor las máquinas moleculares –lo que permitirá desarrollar máquinas biológicas y artificiales a escala nanométrica-, así como para aumentar la eficacia de los antibióticos, que dentro de la maquinaria de la célula podrían comportarse como una especie de llave mecánica capaz de detener determinados procesos celulares dañinos para el organismo.
El diseño de medicamentos basados en la reproducción o simulación sólo de las estructuras estáticas del ribosoma podría compararse con el intento de interceptar en pleno vuelo un misil, si contáramos con la única información de su lugar de lanzamiento y su objetivo. Así, resulta imposible averiguar la trayectoria exacta del misil, del mismo modo que resulta muy difícil conocer los pasos que siguen los ribosomas en sus procesos si no existe una simulación dinámica de éstos.
La reproducción en movimiento de la actividad de los ribosomas ha sido posible gracias a la combinación de diversas ramas de la ciencia: la bioquímica, la informática, la biología molecular, la física, la biología estructural y las ciencias materiales. El resultado permitirá conocer mejor los procesamientos de información a escala nanométrica, tanto artificiales como biológicos. Y es que, de hecho, el ribosoma no es otra cosa que un ordenador a nano escala, un procesador de datos que trabaja para el funcionamiento de la célula.

 Tendencias Científicas
Tendencias Científicas

 La Inteligencia Artificial puede ver a través del espejo
La Inteligencia Artificial puede ver a través del espejo CIENCIA ON LINE
CIENCIA ON LINE